Estudio de UreA, un transportador de urea de una clase exclusiva de hongos y plantas.
Hemos llevado a cabo la caracterización funcional de la permeasa UreA, y el estudio de su regulación a nivel transcripcional. Mediante análisis de la relación estructura-función del transportador, combinando estrategias de mutagénesis y modelado por homología estructural, identificamos residuos clave para su funcionalidad, la unión y translocación del sustrato.
Además del aporte de estos estudios a la biología de hongos y plantas y de esta familia de transportadores en particular, cabría esperar que el mismo sea un punto de partida para el desarrollo de estrategias que permitan un aprovechamiento más eficiente de la urea como fertilizante, o incluso de cultivos con una eficiencia mejorada en cuanto al uso de esta fuente nitrogenada.
Estudio de elementos en cis y factores en trans que regulan el tráfico intracelular de UreA
Un aspecto importante del mantenimiento de la homeostasis celular y las respuestas al estrés es la regulación de la composición de los transportadores en la membrana plasmática (MP), responsables de la absorción y extrusión de nutrientes, así como de otras moléculas pequeñas para las cuales las membranas son impermeables. Buscamos aportar al estudio de la regulación del tráfico intracelular de estos transportadores utilizando como modelo UreA. Hemos identificado elementos en cis presentes en el dominio C-terminal, así como factores en trans (e.g. ubiquitin-ligasa HulA y el adaptador ArtA) que están involucrados en el proceso de degradación de UreA mediado por endocitosis. Asimismo, hemos identificado un posible receptor para proteínas carga (AnErv14) en el retículo endoplásmico, el cual participaría de la regulación de la secreción de UreA hacia la MP.
Un par de codones no óptimos indispensable para la correcta biogénesis de UreA
ureA y sus ortólogos en las otras especies de Aspergillus poseen dos codones no óptimos, conservados que codifican para aminoácidos ubicados exactamente en el límite entre el extremo N-terminal no estructurado y el primer segmento transmembrana. La mutación de estos codones por sus sinónimos óptimos, lleva a una importante disminución en los niveles de UreA, que no se debe a una disminución de los niveles de transcripción del gen ni a un aumento en la degradación de la proteína. Atribuimos entonces los bajos niveles de proteína a un defecto en la síntesis de la misma, que afecta las primeras etapas del proceso, dada la ubicación de los codones. Nuestra hipótesis es que en el mutante sinónimo se ve afectada una pausa traduccional, codificada en el ARNm, necesaria para la correcta interacción con SRP, la partícula de reconocimiento de la señal, que distingue a los polipéptidos nacientes destinados a la membrana del retículo endoplásmico. Actualmente estudiamos la interacción UreA-SRP para verificar esta hipótesis.

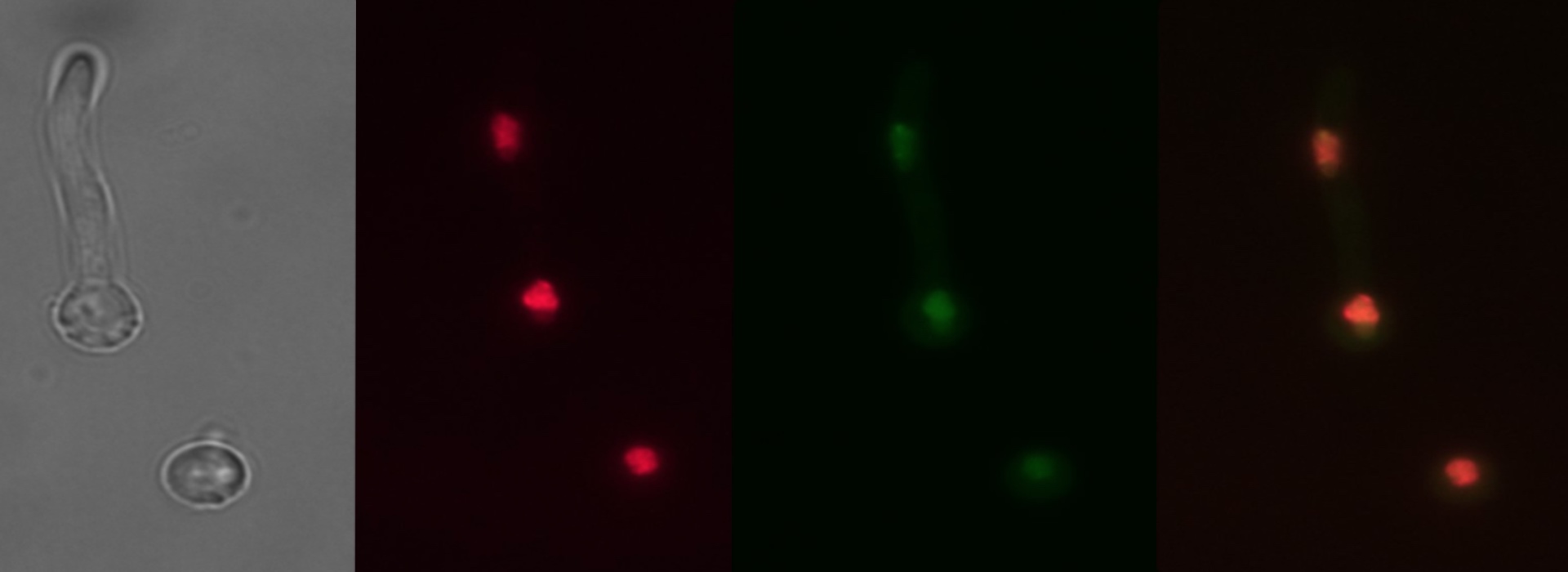
Estructura y función de la cromatina de Aspergillus nidulans
A pesar de la “universalidad” de las características generales de la cromatina, existen diferencias en su estructura y función, incluso dentro de organismos de un mismo género. En el género Aspergillus, hemos detectado diferencias importantes a nivel de las histonas “canónicas” y de sus variantes. Estas diferencias, más allá del interés que revisten desde el punto de vista de la biología celular básica y de la biología de Aspergillus, podrían conducir a la identificación de blancos terapéuticos existen contra infecciones fúngicas, cuya incidencia se ha visto últimamente aumentada de forma dramática especialmente entre pacientes inmuno-comprometidos. En este contexto, estudiamos el rol de H4E, una variante de histona H4 presente exclusivamente en hongos ascomicetos. Esta sería la primera variante de histona H4 descrita hasta el momento.
El EMC de Aspergillus nidulans: caracterización e identificación de clientes
Las proteínas integrales de membrana (PIM) son proteínas transmembrana que participan en una gran variedad de procesos biológicos, como el transporte de solutos (hacia dentro o hacia afuera de la célula) y la transducción de señales. Estas proteínas son codificadas por el ~25% de los genes que codifican para proteínas en todos los organismos y defectos en su función están asociados a varias enfermedades y a la sensibilidad a drogas, lo que destaca su importancia. Se ha identificado un complejo abundante y ampliamente conservado en eucariotas, denominado ER membrane complex (EMC) que ha sido implicado en la biogénesis de estas proteínas. La pérdida de función del EMC se ha asociado a defectos en la homeostasis de colesterol, la formación de autofagosoma, y defectos neurólogicos, entre otros. Por lo tanto, el entender cómo funciona este complejo, así como el identificar sus proteínas clientes (y por ende la selectividad del complejo), es de gran importancia. Es por esto que buscamos contribuir al conocimiento del mismo, mediante su caracterización genética y bioquímica en el hongo ascomicete modelo Aspergillus nidulans.


